胰腺癌(pancreatic cancer)是现今社会病死率较高的消化道恶性肿瘤之一,预后极差,5年生存率不到6%[1-5]。胰腺癌的早期诊断及确诊率不高,目前手术治疗是最佳的治疗方式,但转移和复发率较高,所以找到一个可以判断预后的标记物是很有必要的。相关研究表明,胰腺癌发展的整个过程伴随着多个基因的改变,而细胞周期蛋白依赖性激酶抑制蛋白2A(cyclin-dependent kinase inhibitor 2A,CDKN2A)就是其中之一。CDKN2A是消化道恶性肿瘤患者体内一个突变基因。研究[6]表明CDKN2A在胰腺癌是一个最常见的不良变异基因。在一些胰腺癌患者中,病情的改变已经被证明与CDKN2A突变频率增加相关,最明显的是有胰腺癌发生家族史的患者[7]。所以在高危人群中,一般的变异都足以影响患者的生存率[8]。由于胰腺癌相对较低的发生率和存活率低,其发生的根本原因尚不明确,但有大量研究证明CDKN2A基因对很多肿瘤的发生、发展有着一定程度的影响对肿瘤的发生、发展有着一定的影响[9]。CDKN2A低表达与肿瘤大小、浸润深度、淋巴结转移和TNM分期等临床病理特征有关,可能是一种新的生物标志物,用于预测癌症患者的预后和临床病理特征[10]。CDKN2A主要是产生一种肿瘤抑制蛋白,这是一个细胞周期蛋白依赖性激酶抑制剂[11]。所以,通过认识、了解这个基因的改变特点,有可能为胰腺癌的早期诊断及治疗提供新的思路和方法。
基因拷贝数变异(copy number variation,CNV),是导致人类疾病发生、发展的重要因素之一,主要是因为基因组发生重排,一般指长度为1 kb以上的基因组大片段的拷贝数增加或者减少,主要表现为亚显微水平的缺失和重复。CDKN2A基因在胰腺癌的CNV程度较高。本研究通过TCGA及GTEx等数据库研究胰腺癌中CDKN2A基因CNV对其表达和预后的影响,为进一步研究CDKN2A相关信号通路在胰腺癌中的作用机制提供新的思路。
1 材料与方法
1.1 研究对象
癌症基因组图谱(TCGA)数据库里有关胰腺癌数据库的临床资料,cBioPortal是TCGA的一个可视化在线数据库,可以通过在cBioPortal(http://www.cbioportal.org)中“Pancreatic cancer(TCGA,Provisional)186 samples下载临床数据。CNV、mRNA表达RNA Seq V2可在子数据库分析,同时使用GEO等数据库。
1.2 数据分析
分析统计186例胰腺癌样本中包含CDKN2A基因CNV的样本以及未包含CDKN2A基因CNV的样本的各项临床参数,从TCGA等数据库收集胰腺癌数据及其临床资料,研究CDKN2A基因拷贝数缺失患者的临床病理特征,利用TCGA数据库CNV与RNA Seq V2数据分析CDKN2A的CNV与其mRNA的表达水平的相关性。分析CDKN2A基因CNV对GGA2、MTAP、HOXC6、SLC2A1表达的影响。通过比对TCGA数据库及GTEx数据库研究CDKN2A基因在正常人群与胰腺癌患者间的表达差异、CDKN2A基因CNV与胰腺癌患者预后的相关性;使用DAVID在线分析网站,通过对CDKN2A及其共表达基因进行基因集富集分析,得到CDKN2A基因富集通路。
1.3 统计学处理
生存分析使用Kaplan-Meier和Log-rank检验,P<0.05为相关结果有差异,且有统计学意义。其它基因表达与CNV的相关性以及共表达蛋白的相关性使用Spearman和Pearson检验,r>0.3认为有统计学相关。功能分析采用Fisher检验,P<0.05为功能富集有统计学差异。
2 结 果
2.1 CDKN2A基因CNV以及与其mRNA表达的相关性
胰腺癌中C D K N 2 A基因C N V情况见图1。CDKN2A基因CNV与mRNA表达呈正相关(Pearson:r=0.102,Spearman:r=0.257)(图2)。随着CDKN2A基因CNV程度增高,其表达也随之升高。
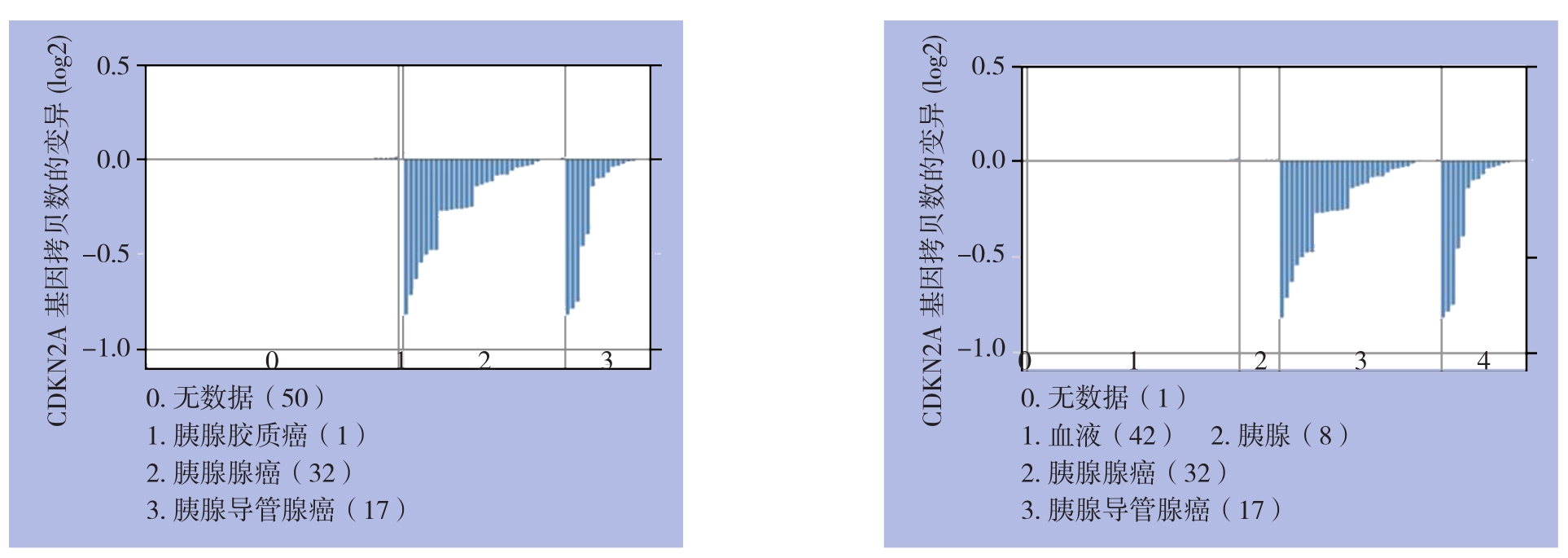
图1 胰腺癌中CDKN2A基因CNV情况
Figure 1 CDKN2A gene CNV in pancreatic cancers
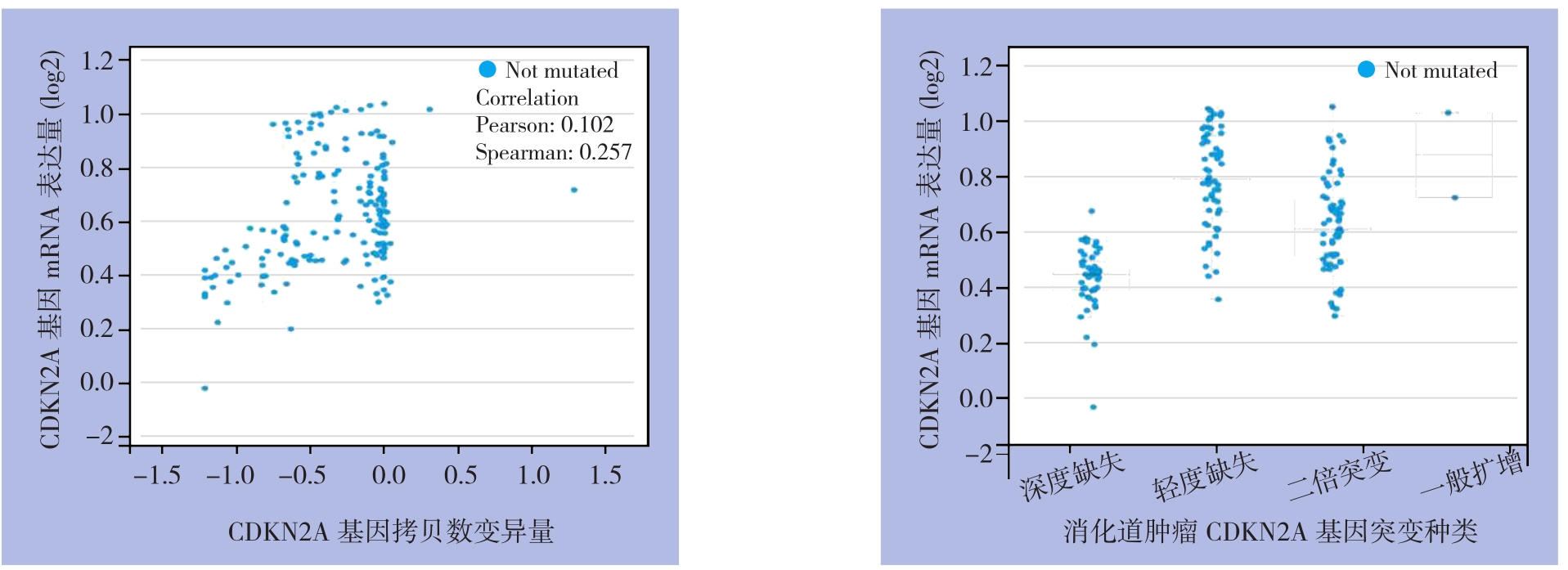
图2 CDKN2A基因CNV与其mRNA表达的相关性
Figure 2 Correlation of CDKN2A gene CNV to its mRNA expression
2.2 CDKN2A基 因 CNV对 GGA2、MTAP、HOXC6、SLC2A1表达的影响
将1 8 6例研究样本根据有无C D K N 2 A基因CNV分为变异组与非变异组,结合TCGA数据库RNA Seq V2数据分析CDKN2A基因CNV对GGA2、MTAP、HOXC6、SLC2A1表达的影响,使用Student t-test检验。与非变异组相比,CDKN2A基因CNV组中HOXC6、SLC2A1 mRNA表达水平明显升高,GGA2、MTAP mRNA表达水平明显降低(均P<0.05)(图3)。通过相关性研究可以发现,CDKN2A基因与MTAP为正相关(Pearson:r=0.42,Spearman:r=0.55,P<0.05),与其他三者相关程度较低(图4)。
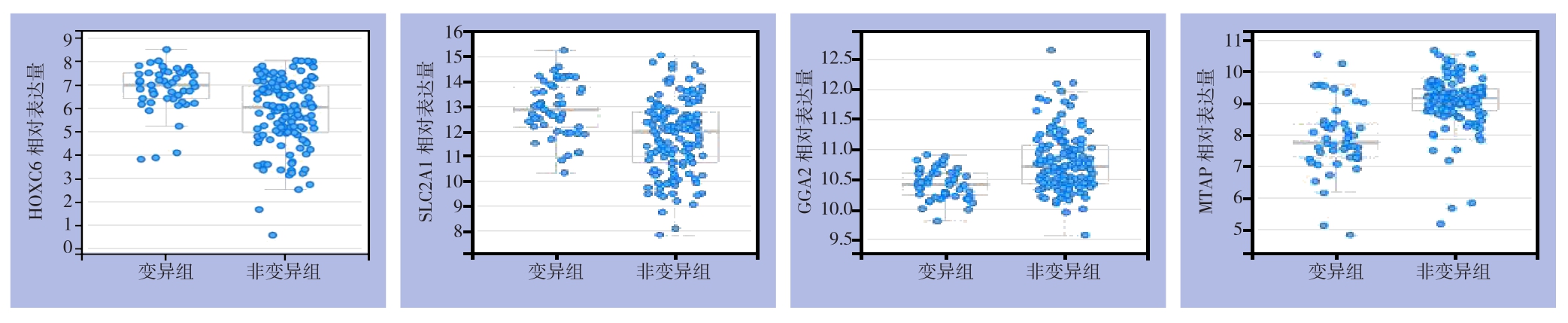
图3 变异组与非变异组各基因mRNA表达情况
Figure 3 The mRNA expressions of each studied gene in altered group and unaltered group
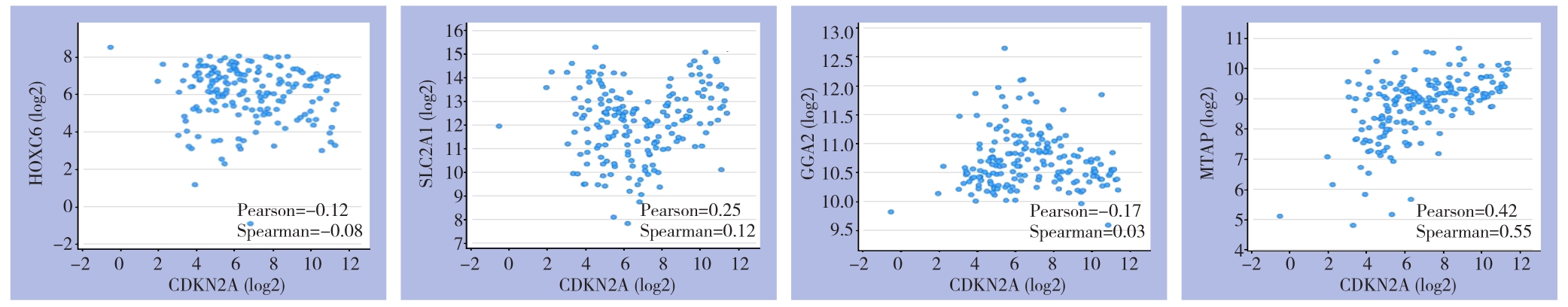
图4 CDKN2A与各个基因相关性散点图
Figure 4 Scatter plots showing correlations of CDKN2A with related genes
2.3 CDKN2A基因在胰腺癌与正常组织中表达的差异
通过比对TCGA数据库179例胰腺癌样本及GTEx数据库171例正常胰腺样本发现,CDKN2A基因在胰腺癌肿瘤组织中表达明显高于正常胰腺组织(P<0.05)(图5)。
2.4 CDKN2A基因CNV与胰腺癌患者预后的相关性
携带CDKN2A基因CNV的胰腺癌患者的总生存率(OS)明显低于不携带该基因CNV的胰腺癌患者(P=0.0175),CDKN2A基因CNV患者的无瘤生存率也明显低于不携带该基因CNV的患者(P=0.0368)(图6)。
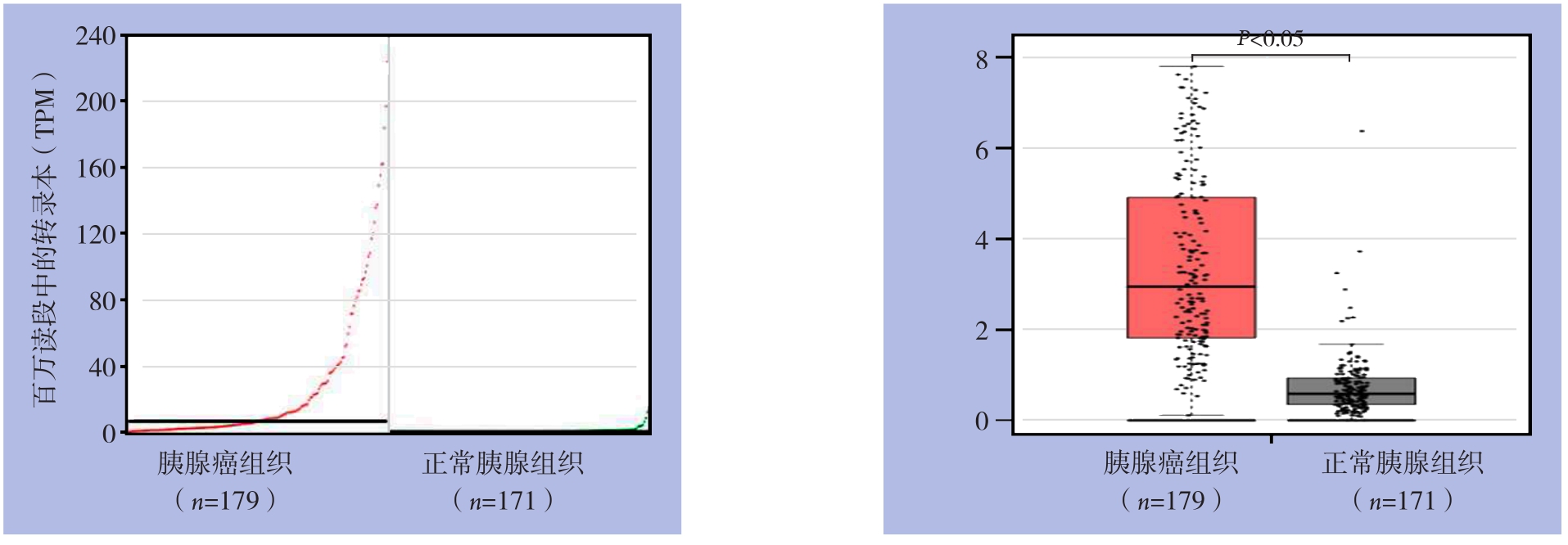
图5 CDKN2A基因在胰腺癌和正常胰腺组织中的表达
Figure 5 CDKN2A gene expressions in pancreatic cancer and normal pancreatic tissues
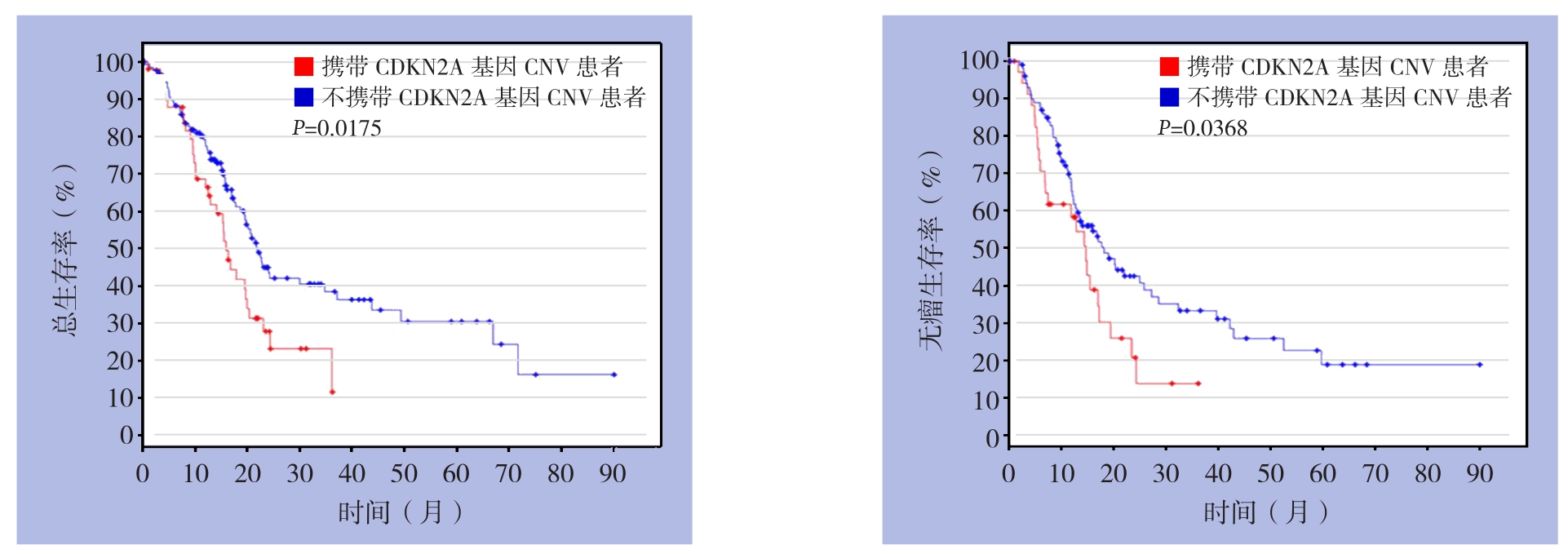
图6 CDKN2A基因CNV与胰腺癌患者预后的相关性
Figure 6 Correlation between CDKN2A gene CNV and prognosis in patients with pancreatic cancer
2.5 CDKN2A的功能基因富集分析
通过对CDKN2A基因相关共表达基因分析发现,CDKN2A基因CNV与一些基因表达水平存在明显的关联(Pearson≥0.3或Spearman≥0.3)(表1)。通过对包括CDKN2A相关表达KEGG功能富集分析发现(表2),这些共表达基因的功能主要集中于一些细胞周期检查点的信号通路。提示CDKN2A相关共表达基因通过细胞周期通路调节肿瘤的发生发展。
表1 CDKN2A基因相关共表达基因分析
Table 1 Analysis of the coexpression genes related to CDKN2A gene
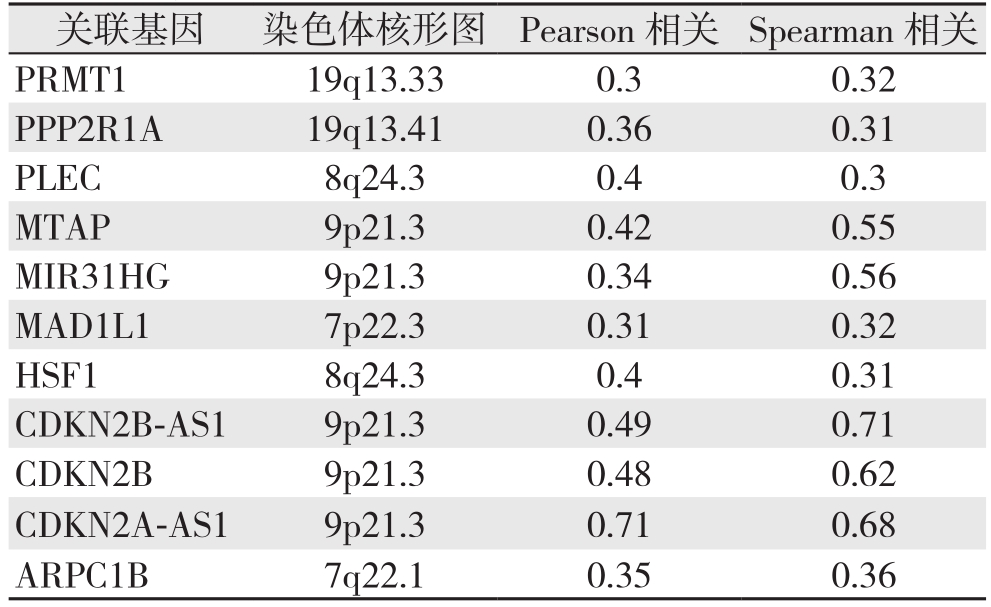
关联基因 染色体核形图 Pearson相关 Spearman相关PRMT1 19q13.33 0.3 0.32 PPP2R1A 19q13.41 0.36 0.31 PLEC 8q24.3 0.4 0.3 MTAP 9p21.3 0.42 0.55 MIR31HG 9p21.3 0.34 0.56 MAD1L1 7p22.3 0.31 0.32 HSF1 8q24.3 0.4 0.31 CDKN2B-AS1 9p21.3 0.49 0.71 CDKN2B 9p21.3 0.48 0.62 CDKN2A-AS1 9p21.3 0.71 0.68 ARPC1B 7q22.1 0.35 0.36
表2 CDKN2A的功能基因集富集分析
Table 2 Enrichment analysis of CDKN2A functional genes
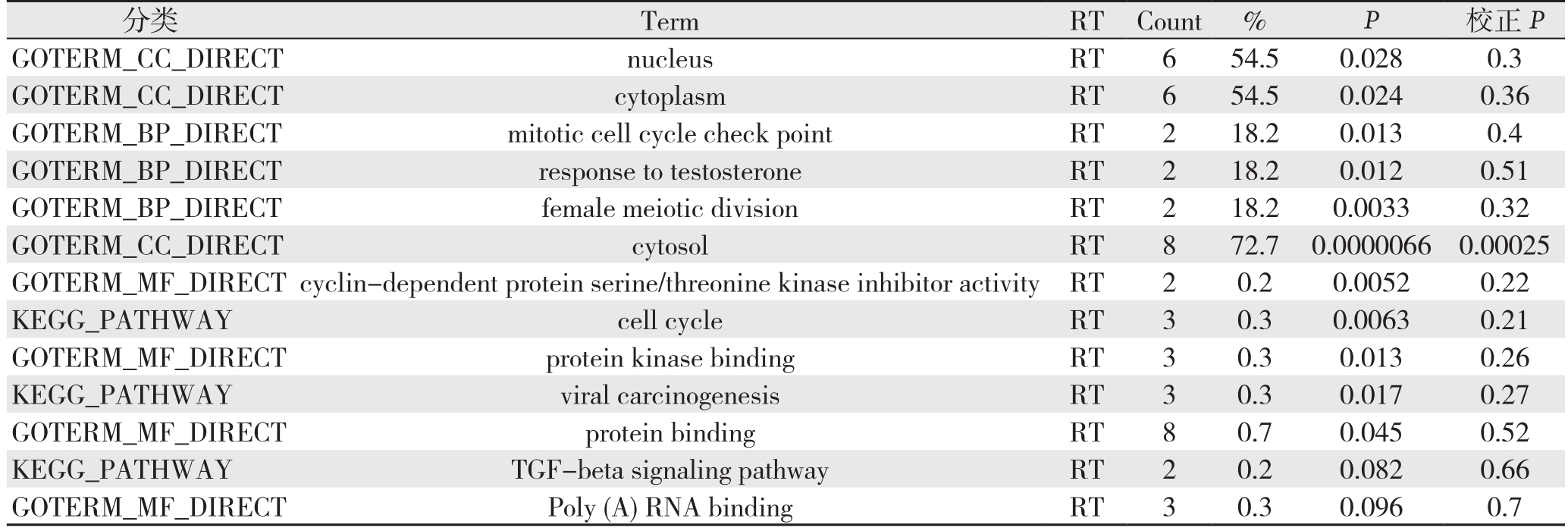
分类 Term RT Count % P 校正P GOTERM_CC_DIRECT nucleus RT 6 54.5 0.028 0.3 GOTERM_CC_DIRECT cytoplasm RT 6 54.5 0.024 0.36 GOTERM_BP_DIRECT mitotic cell cycle check point RT 2 18.2 0.013 0.4 GOTERM_BP_DIRECT response to testosterone RT 2 18.2 0.012 0.51 GOTERM_BP_DIRECT female meiotic division RT 2 18.2 0.0033 0.32 GOTERM_CC_DIRECT cytosol RT 8 72.7 0.0000066 0.00025 GOTERM_MF_DIRECT cyclin-dependent protein serine/threonine kinase inhibitor activity RT 2 0.2 0.0052 0.22 KEGG_PATHWAY cell cycle RT 3 0.3 0.0063 0.21 GOTERM_MF_DIRECT protein kinase binding RT 3 0.3 0.013 0.26 KEGG_PATHWAY viral carcinogenesis RT 3 0.3 0.017 0.27 GOTERM_MF_DIRECT protein binding RT 8 0.7 0.045 0.52 KEGG_PATHWAY TGF-beta signaling pathway RT 2 0.2 0.082 0.66 GOTERM_MF_DIRECT Poly (A) RNA binding RT 3 0.3 0.096 0.7
3 讨 论
胰腺癌作为恶性程度较高的消化道肿瘤之一,一旦发现,大多达到进展期,放化疗等综合治疗对患者生存时间的改善甚微,其治疗的首选方案为手术,但长期的临床实践表明,对于胰腺癌单纯手术的治疗效果是有限的[12]。因此,早期发现并诊断胰腺癌成为目前迫在眉睫的问题。通过对胰腺癌基因的调查,发现了以前的基因研究尚不完善[13]。在过去的几十年里,有一些分子标志物被研究出来,作为胰腺癌早期诊断的基因标志物[14]。CA19-9是一种黏蛋白类物质,因为其诊断胰腺癌的敏感性和特异性分别达到80.0%和86.0%,所以被广泛应用于胰腺癌的诊断[15]。但CA19-9还存在一些不足,其在胰腺良性疾病和非胰腺肿瘤患者的检测中也有升高,如在胰腺炎、胆囊炎、胃癌、结直肠癌中等等[16]。而且,尽管CA19-9已经使用几十年,但仍缺乏定量诊断标准[17]。CEA则是一种酸性糖蛋白,可用于胰腺癌的辅助诊断,但是因其在多种癌症,如乳腺癌、消化道肿瘤、肺癌等癌症表达都有所升高,所以特异性并不高[18-21]。CDKN2A基因在许多肿瘤的发生、发展中有着不同的改变,使它对于肿瘤的密切关系与日俱增。胰腺癌患者存在CDKN2A基因突变频率增加的情况[22]。并且胰腺癌中CDKN2A大多数的变异是基因缺失[23],CDKN2A基因主要是通过肿瘤细胞周期抑制剂调控肿瘤进展[24-25]。所以,通过认识、了解这些基因的改变特点,有可能为胰腺癌的早期诊断及治疗提供新的思路和方法。
本研究结果特异性地证实了在胰腺癌中,CDKN2A基因CNV是一种预后较差的因素。利用TCGA数据库CNV、RNA Seq V2数据分析CDKN2A的CNV与其mRNA的表达水平的相关性。CDKN2A基因拷贝数的变异与mRNA表达呈明显正相关(Pearson:r=0.102,Spearman:r=0.257)。表明随着CDKN2A基因CNV程度增高,其mRNA表达也随之升高。结合 TCGA 数据库RNA Seq V2数据分析 CDKN2A基因CNV对GGA2、MTAP、HOXC6、SLC2A1表达的影响。CDKN2A基因CNV组中HOXC6、SLC2A1 mRNA表达水平明显升高。GGA2、MTAP mRNA表达水平明显降低(P<0.05)。而通过相关性研究可以发现,CDKN2A基因与MTAP为明显正相关(Pearson:r=0.42,Spearman:r=0.55,均P<0.05),与其他三者相关程度较低,可以得出CDKN2A很有可能使MTAP表达下降。
CDKN2A基因在肿瘤和正常组织中表达的差异,通过比对TCGA数据库179例肿瘤样本及GTEx数据库171例正常样本发现,CDKN2A基因在胰腺癌肿瘤组织中表达显著高于正常组织。结合TCGA数据库183例样本中基因表达与临床资料分析发现,CDKN2A基因在各临床分期中表达无显著差异。CDKN2A基因CNV水平与OS显著相关(P=0.0175),携带CDKN2A基因CNV的胰腺癌患者预后明显差于不携带该基因CNV的胰腺癌患者。另外CDKN2A基因CNV患者的无瘤生存期显著短于不携带该基因CNV的患者(P=0.0368)。表明CDKN2A变异提示预后不良,患者总生存期和无瘤生存期显著缩短。通过对CDKN2A及其共表达基因KEGG 功能富集分析,发现这些共表达基因的功能主要集中于一些细胞周期的信号通路,提示CDKN2A相关共表达基因通过细胞周期通路调节肿瘤的发生发展。
综上所述,本研究依托TCGA数据库,对胰腺癌患者进行大样本量的数据分析和临床资料对比分析,探讨CDKN2A CNV对患者预后的影响。通过了解基因CNV的程度,可确定CDKN2A基因可作为胰腺癌预后评估的重要分子标志物。
[1]Tang B,Li Y,Qi G,et al.Clinicopathological Significance of CDKN2A Promoter Hypermethylation Frequency with Pancreatic Cancer[J].Sci Rep,2015,5:13563.doi: 10.1038/srep13563.
[2]Kim CB,Ahmed S,Hsueh EC.胰腺癌的手术治疗现状[J].中国普通外科杂志,2013,22(9):1105–1113.doi:10.7659/j.issn.1005–6947.2013.09.001.Kim CB,Ahmed S,Hsueh EC.Current surgical management of pancreatic cancer[J].Chinese Journal of General Surgery,2013,22(9):1105–1113.doi:10.7659/j.issn.1005–6947.2013.09.001.
[3]汤首俊,朱世凯.长链非编码RNA在胰腺癌中的研究进展[J].中国普通外科杂志,2016,25(3):443–447.doi:10.3978/j.issn.1005–6947.2016.03.024.Tang SJ,Zhu SK.Long non-coding RNAs in pancreatic cancer:recent advances[J].Chinese Journal of General Surgery,2016,25(3):443–447.doi:10.3978/j.issn.1005–6947.2016.03.024.
[4]杨尹默.胰腺癌外科治疗的现状、存在问题与展望[J].中国普通外科杂志,2016,25(9):1231–1235.doi:10.3978/j.issn.1005–6947.2016.09.001.Yang YM.Surgical managements of pancreatic cancer:current status and future directions[J].Chinese Journal of General Surgery,2016,25(9):1231–1235.doi:10.3978/j.issn.1005–6947.2016.09.001.
[5]钟志惟,殷香保.胰腺癌的分子靶向治疗研究进展[J].中国普通外科杂志,2016,25(9):1351–1356.doi:10.3978/j.issn.1005–6947.2016.09.022.Zhong ZW,Yin XB.Research progress in molecular targeted therapy for pancreatic cancer[J].Chinese Journal of General Surgery,2016,25(9):1351–1356.doi:10.3978/j.issn.1005–6947.2016.09.022.
[6]Campa D,Pastore M,Gentiluomo M,et al.Functional single nucleotide polymorphisms within the cyclin-dependent kinase inhibitor 2A/2B region affect pancreatic cancer risk[J].Oncotarget,2016,7(35):57011–57020.doi: 10.18632/oncotarget.10935.
[7]Yang XR,Rotunno M,Xiao Y,et al.Multiple rare variants in highrisk pancreatic cancer-related genes may increase risk for pancreatic cancer in a subset of patients with and without germline CDKN2A mutations[J].Hum Genet,2016,135(11):1241–1249.doi: 10.1007/s00439–016–1715–1.
[8]Wang Y,Zhang Y,Yang J,et al.Genomic sequencing of key genes in mouse pancreatic cancer cells[J].Curr Mol Med,2012,12(3):331–341.
[9]McWilliams RR,Wieben ED,Rabe KG,et al.Prevalence of CDKN2A mutations in pancreatic cancer patients: implications for genetic counseling[J].Eur J Hum Genet,2011,19(4):472–478.doi:10.1038/ejhg.2010.198.
[10]Childs EJ,Chaffee KG,Gallinger S,et al.Association of Common Susceptibility Variants of Pancreatic Cancer in Higher-Risk Patients:A PACGENE Study[J].Cancer Epidemiol Biomarkers Prev,2016,25(7):1185–1191.doi:10.1158/1055–9965.EPI-15–1217.
[11]Potjer TP,van der Stoep N,Houwing-Duistermaat JJ,et al.Pancreatic cancer-associated gene polymorphisms in a nation-wide cohort of p16-Leiden germline mutation carriers; a case-control study[J].BMC Res Notes,2015,8:264.doi: 10.1186/s13104–015–1235–4.
[12]黄东胜,杨坡,刘军伟,等.ADAM9在胰腺癌发生发展中的作用研究[J].中华普通外科杂志,2010,25(11):931–932.doi.10.3760/cma.j.issn.1007–631X.2010.11.027.Huang DS,Yang P,Liu JW,et al.Role of ADAM9 in occurrence and development of pancreatic cancer[J].Zhong Hua Pu Tong Wai Ke Za Zhi,2010,25(11):931–932.doi.10.3760/cma.j.issn.1007–631X.2010.11.027.
[13]Shain AH,Salari K,Giacomini CP,et al.Integrative genomic and functional profiling of the pancreatic cancer genome[J].BMC Genomics,2013,14:624.doi:10.1186/1471–2164–14–624.
[14]赵建国,廖泉,赵玉沛.胰腺癌分子标志物研究进展[J].中华普外科手术学杂志:电子版,2013,7(3):224–226.doi:10.3877/cma.j.issn.1674–3946.2013.03.060.Zhao JG,Liao Q,Zhao YP.The development of molecular markers for pancreatic cancer[J].Chinese Journal of Operative Procedures of General Surgery: Electronic Version,2013,7(3):224–226.doi:10.3877/j.issn.1674–3946.2013.03.060.
[15]Duffy MJ,Sturgeon C,Lamerz R,et al.Tumor markers in pancreatic cancer: a European Group on Tumor Markers(EGTM)status report[J].Ann Oncol,2010,21(3):441–447.doi: 10.1093/annonc/mdp332.
[16]Kulasingam V,Diamandis EP.Strategies for discovering novel cancer biomarkers through utilization of emerging technologies[J].Nat Clin Pract Oncol,2008,5(10):588–599.doi: 10.1038/ncponc1187.
[17]La'ulu SL,Roberts WL.Performance characteristics of five automated CA 19–9 assays[J].Am J Clin Pathol,2007,127(3):436–440.doi:10.1309/H52VET3M6P7GYWG1.
[18]杨泽安.CEA、CA125及CA153在乳腺癌中的检测价值[J].湖南师范大学学报:医学版,2016,13(3):83–85.Yang ZA.The detective value of CEA,CA125and CA153 in mammary cancer[J].Journal of Hunan Normal University: Medical Science,2016,13(3):83–85.
[19]蒲瑞雪,赵芹.癌胚抗原(CEA)与糖类抗原(CA125、CA153)对乳腺癌的诊断价值[J].医学检验与临床,2016,27(1): 60–61.doi:10.3969/j.issn.1673–5013.2016.01.023.Pu RX,Zhao Q.Diagnostic value of carcino-embryonic antigen(CEA) and carbohydrate antigens (CA125,CA153) for breast cancer[J].Medical Laboratory Science and Clinics,2016,27(1):60–61.doi:10.3969/j.issn.1673–5013.2016.01.023.
[20]傅丽敏,程雪,张小薇.血清肿瘤标记物癌胚抗原、CA153和CA125检测对乳腺癌的临床诊断价值[J].中国现代医生,2016,54(20):104–107.Fu LM,Cheng X,Zhang XW.Significance of serum tumor markers CEA and CAl53,CAl25 in breast cancer diagnosis[J].Chinese Modern Doctor,2016,54(20):104–107.
[21]陈艺文.CA153、CA125及CEA联合检测对乳腺癌诊断的意义[J].农垦医学,2012,34(5):401–403.doi:10.3969/j.issn.1008–1127.2012.05.005.Chen YW.The value of combined assay of CA153,CA125 and CEA in the diagnosis of breast cancer[J].Journal of Nongken Medicine,2012,34(5):401–403.doi: 10.3969/j.issn.1008–1127.2012.05.005.
[22]Yang XR,Jessop L,Myers T,et al.Lack of germline PALB2 mutations in melanoma-prone families with CDKN2A mutations and pancreatic cancer[J].Fam cancer,2011,10(3):545–548.doi:10.1007/s10689–011–9447–9.
[23]Norris AL,Kamiyama H,Makohon-Moore A,et al.Transflip mutations produce deletions in pancreatic cancer[J].Genes Chromosomes Cancer,2015,54(8):472–481.doi: 10.1002/gcc.22258.
[24]Helgadottir H,Höiom V,Jönsson G,et al.High risk of tobaccorelated cancers in CDKN2A mutation-positive melanoma families[J].J Med Genet,2014,51(8):545–552.doi:10.1136/jmedgenet-2014–102320.
[25]Hélias-Rodzewicz Z,Lourenco N,Bakari M,et al.CDKN2A depletion causes aneuploidy and enhances cell proliferation in nonimmortalized normal human cells[J].Cancer Invest,2018,23:1–11.doi: 10.1080/07357907.2018.1491588.