结直肠癌(colorectal cancer,CRC)患者中Lynch综合征约占2%~3%[1-2],其主要特点是发生家族性结直肠肿瘤或肠外肿瘤。DNA错配修复基因(mismatch repair,MMR)发生种系突变是Lynch综合征患者发病的遗传学基础,MLH1、MSH2、MSH6和PMS2是最常见发生突变的4个错配修复基因。MMR的主要功能是纠正DNA复制时单个核苷酸的错误配对,其发生突变引起微卫星不稳定(microsatellite instability,MSI)[3],DNA复制不能正常进行或遗传信息发生错误传递。应用免疫组化方法检测结直肠患者肿瘤组织标本的4个错配修复基因,以及配合MLH1启动子甲基化和BRAF突变分析已逐渐推荐应用于临床散在性MMR缺失的筛查[4]。PMS2参与DNA错配修复并且在肿瘤易感性中起作用,其编码的蛋白质产物与MLH1结合形成异源二聚体hMutL-a(hMLH1/hPMS2),识别错配位点,参与错配修复[5]。本研究旨在分析结直肠癌肿瘤组织PMS2蛋白的表达状态及其与临床病理参数的关系及PMS2蛋白表达状态与血清CEA、CA19-9、CA125水平的关系及相关性,期以为临床诊治提供参考。
1 材料与方法
1.1 材料
收集中南大学湘雅医院普通外科2017年5月—2018年7月确诊的结直肠癌切除手术肿瘤组织标本1 023例及相应患者的血清肿瘤标记物CEA、CA19-9、CA125,其中结肠癌肿瘤组织520例,直肠癌肿瘤组织503例。所有患者中男595例,女428例;年龄为25~89岁,平均年龄为59.4岁。
1.2 方法
所有标本均经10%的中性福尔马林液固定,石蜡包埋。HE染色切片,经2名以上病理医师诊断。免疫组化使用北京中杉金桥PV6000型免疫组化试剂盒,步骤如下:石蜡切片脱蜡、水化后PBS冲洗,EDTA高压加热进行抗原修复,过氧化物酶阻断剂封闭内源性过氧化酶活性,加入一抗,37 ℃孵育1 h,加酶标二抗37 ℃孵育30 min,加DAB显色液显色约4 min,显微镜下观察控制显色程度,苏木素复染,常规脱水,透明,封片。PMS2抗体购于福州迈新生物技术开发有限公司。PMS2蛋白定位于细胞核。以肿瘤周围的正常上皮细胞、肿瘤内浸润的淋巴细胞、间质细胞作为内对照,如内对照阳性则纳入研究,如内对照阴性则剔除。PMS2蛋白阴性表达即肿瘤细胞核与内对照相比无着色(图1A),PMS2蛋白阳性表达即肿瘤细胞核出现与内对照一致的着色(图1B)。

图1 PMS2蛋白在结直肠癌肿瘤组织中的表达(SP,标尺100 μm) A:PMS2蛋白在结直肠癌肿瘤组织中阴性表达;B:PMS2蛋白在结直肠癌肿瘤组织中阳性表达
1.3 统计学处理
采用GraphPad Prism 5软件分析,计数资料率的比较采用χ2检验、Fisher确切概率法,相关性分析采用Spearman等级相关检验,P<0.05为差异有统计学意义。
2 结 果
2.1 结肠癌肿瘤组织和直肠癌肿瘤组织的PMS2蛋白的表达及关系
结肠癌肿瘤组织中,PMS2蛋白阴性表达率为14.0%(73/520),直肠癌肿瘤组织的PMS2蛋白阴性表达率为2.9%(13/503),结肠癌肿瘤组织的PMS2蛋白阴性表达率明显高于直肠癌(P<0.000 1)(表1)。
表1 结肠癌与直肠癌肿瘤组织的PMS2蛋白表达状态比较

部位 n PMS2(-) PMS2(+) χ2 P结肠 520 73(14.0) 447(86.0) 43.56 <0.000 1直肠 503 13(2.6) 490(97.4)
2.2 结肠癌肿瘤组织中PMS2蛋白表达与临床病理参数的关系
结肠癌肿瘤组织PMS2蛋白的阴性表达者与阳性表达者的周围神经侵犯阳性率分别为4.1%(3/73)、14.8%(66/447),阴性表达者周围神经侵犯阳性率明显低于阳性表达者(P=0.009 1);结肠癌肿瘤组织PMS2蛋白的阴性表达者与阳性表达者的肿瘤最大径≥5 cm的比例分别为69.9%(51/73)、47.2%(211/447),阴性表达者肿瘤最大径≥5 cm的比例明显高于阳性表达者(P=0.000 3);结肠癌肿瘤组织PMS2蛋白的阴性表达者的肿瘤细胞低分化的比例明显高于PMS2蛋白的阳性表达者(P=0.000 6);两者在性别、年龄、脉管侵犯阳性率、系膜内淋巴转移发生率及T分期均无统计学差异(P>0.05)(表2)。
2.3 结肠癌肿瘤组织PMS2蛋白表达状态与血清CEA、CA19-9、CA125水平的关系及其相关性
结肠癌肿瘤组织的PMS2蛋白的阴性表达和阳性表达者血清CEA、CA19-9、CA125水平无统计学差异;且两者无明显相关性(P>0.05)(表3)。
表2 结肠癌肿瘤组织PMS2蛋白的表达状态与各临床病理参数的关系

注:1)采用Fisher确切概率法
images/BZ_135_213_404_2268_453.pngimages/BZ_135_213_601_2268_1045.png部位 神经侵犯左半结肠 296 39(13.2) 257(86.8) 0.4239 0.515 0 是 69 3(4.3) 66(95.7) — 0.009 11)右半结肠 224 34(15.2) 190(84.8) 否 451 70(15.5) 381(84.5)脉管侵犯是128 17(13.3) 111(86.7) 0.08067 0.776 4否 392 56(14.3) 336(85.7)
表3 结肠癌肿瘤组织PMS2蛋白表达状态与血清CEA、CA19-9、CA125水平的关系及其相关性
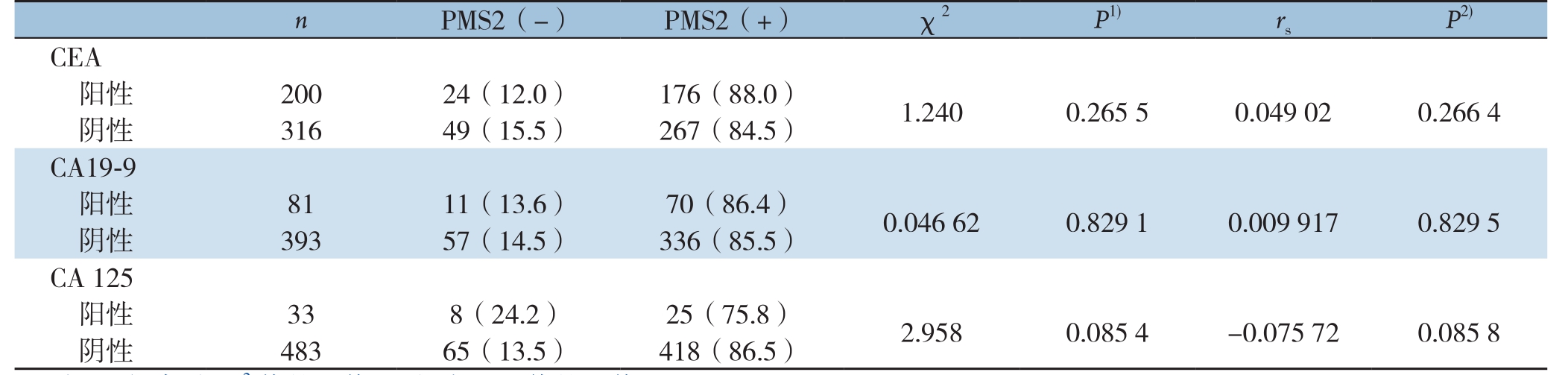
注:1)表示χ2值的P值;2)表示rs值的P值
CEA阳性 200 24(12.0) 176(88.0) 1.240 0.265 5 0.049 02 0.266 4阴性 316 49(15.5) 267(84.5)CA 125阳性 33 8(24.2) 25(75.8) 2.958 0.085 4 -0.075 72 0.085 8阴性 483 65(13.5) 418(86.5)images/BZ_135_213_1349_2269_1398.pngimages/BZ_135_213_1546_2269_1694.png
2.4 直肠癌肿瘤组织中PMS2蛋白表达与临床病理参数的关系
直肠癌肿瘤组织PMS2蛋白的阴性表达者与阳性表达者T3+4分期比率分别为 100.0%(13/13)、72.7%(356/490),阴性表达者T3+T4分期比率明显高于阳性表达者(P=0.024 6);两者在性别、年龄、肿瘤大小、脉管侵犯阳性率、神经侵犯率、系膜内淋巴转移发生率及肿瘤细胞低分化比例均无统计学差异(P>0.05)(表4)。
表4 直肠癌肿瘤组织PMS2蛋白的表达状态与各临床病理参数的关系

注:1)采用Fisher确切概率法
images/BZ_135_213_2447_2266_2496.pngimages/BZ_135_213_2644_2266_3098.png
2.5 直肠癌肿瘤组织PMS2蛋白表达状态与血清CEA、CA19-9、CA125水平的关系及其相关性
直肠癌肿瘤组织的PMS2蛋白的阴性表达者与阳性表达者的血清CA19-9水平无统计学差异(P>0.05),但PMS2蛋白表达和血清CA19-9水平呈负相关(r=-0.162 3,P<0.01);两者在血清CEA、CA125水平均无统计学差异,且两者无明显相关性(P>0.05)(表5)。
表5 直肠癌肿瘤组织PMS2蛋白表达状态与血清CEA、CA19-9、CA125水平的关系及其相关性
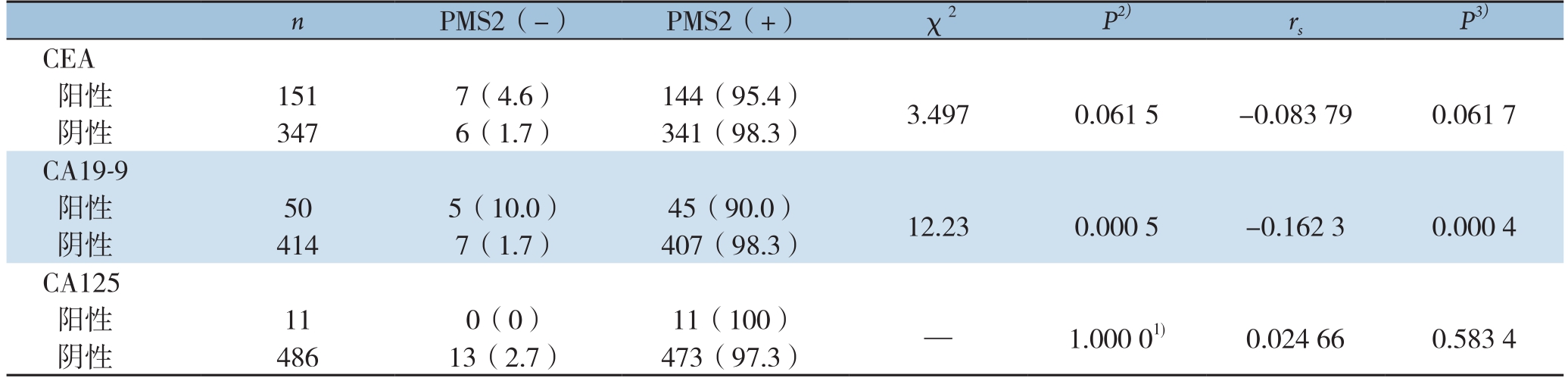
注:1)采用Fisher确切概率法;2)χ2值的P值;3)rs值的P值
n PMS2(-) PMS2(+) χ2 P2) rs P3)CEA阳性 151 7(4.6) 144(95.4) 3.497 0.061 5 -0.083 79 0.061 7阴性 347 6(1.7) 341(98.3)CA19-9阳性 50 5(10.0) 45(90.0) 12.23 0.000 5 -0.162 3 0.000 4阴性 414 7(1.7) 407(98.3)CA125阳性 11 0(0) 11(100) — 1.000 01) 0.024 66 0.583 4阴性 486 13(2.7) 473(97.3)
3 讨 论
DNA错配修复系统是指在含有错配碱基的DNA分子中,使核苷酸序列恢复正常的修复方式。DNA错配修复系统是一个严格的校对者,鉴别及纠正复制时错配的DNA碱基,保证基因组的完整性和稳定性,避免遗传物质发生突变。错配修复基因通过编码错配修复蛋白修正这些错误。错配修复基因突变导致其蛋白表达产物产生缺陷而失去错配修复功能,从而导致DNA复制错误,引起细胞增殖及异常分化增加,最终导致多种肿瘤的发生[6]。MLH1、MSH2、MSH6、PMS2是DNA错配修复系统的主导蛋白[7-8],4种蛋白均正常表达即为错配修复正常(pMMR),至少有1项表达缺失为错配修复蛋白表达缺失(dMMR)。PMS2基因是错配修复系统中的一个较为重要的基因,其在结直肠癌中有一定比例的阴性表达[9]。
P M S 2基因位于染色体7 p 2 2,c D N A有2586bp。PMS2基因突变最初描述于遗传性非息肉病性结肠直肠癌(Lynch综合征)[10]。PMS2参与DNA错配修复并且在肿瘤易感性中起作用,其编码的蛋白质产物与MLH1结合形成异源二聚体hMutL-a(hMLH1/hPMS2),识别错配位点,参与错配修复[5]。有证据表明PMS2蛋白和其它一些错配修复蛋白是多功能的,可以影响细胞周期进程和细胞凋亡[11]。
目前的研究热点多集中在MMR表达水平在左半结肠癌、右半结肠癌与直肠癌之间的差异[12]。本研究结果显示结肠癌肿瘤组织中PMS2蛋白表达阴性病例的比例明显高于直肠癌肿瘤组织。结肠与直肠的胚胎发育和生理环境及功能存在差异[13],肿瘤组织中PMS2蛋白表达在结肠与直肠有明显差异可能与之有关。有文献报道,dMMR的肿瘤组织常有较差的分化[14],但有较好的预后[15-17],可能与对化疗反应不同有关[14]。但少见单独PMS2蛋白表达水平与结直肠癌预后关系报道,Ten Broeke等[18]报道PMS2相关Lynch综合征患者与其它错配修复基因缺陷所致Lynch综合征不同,PMS2相关结直肠癌肿瘤组织的KRAS突变率高于MLH1和MSH2。本研究中PMS2蛋白阴性表达者T3+T4期比例和肿瘤细胞分化程度差比例明显高于阳性表达者,与文献[6]报道dMMR肿瘤组织有较差分化相符。本研究结果显示直肠癌肿瘤组织的PMS2蛋白阳性表达者周围神经侵犯率明显高于阴性表达者,肿瘤组织侵犯神经,提示常更易复发和转移。最新的文献[19-20]报道,相比于结直肠癌的pMMR肿瘤组织,dMMR的肿瘤组织的PD-L1表达率更高,微卫星高不稳定的肿瘤组织的PD-L1表达率要显著高于微卫星稳定的患者[21],提示dMMR及微卫星高不稳定的肿瘤组织可能对抗PD-1/L1免疫治疗有更多的获益[20]。本研究显示,大部分直肠癌肿瘤组织标本的PMS2蛋白阳性表达,大部分相应患者血清CA19-9表达阴性,PMS2蛋白的表达与CA19-9水平呈负相关。
本研究局限性为近1年多的患者(相当一部分未满1年),因此需获取长期随访结果有利于进一步了解预后情况。本研究为回顾性研究,在此期间主要收集了1 023例结直肠癌的病例资料,同期的腺瘤非癌病例有数万例,并未行腺瘤组织样本的MMR表达分析。
总之,PMS2蛋白表达状态与结直肠癌临床病理因素密切相关,可能对于指导临床诊疗和预后分析有一定价值。
[1]Carethers JM, Stoffel EM.Lynch syndrome and Lynch syndrome mimics: The growing complex landscape of hereditary colon cance[J].World J Gastroenterol, 2015, 21(31):9253-9261.doi:10.3748/wjg.v21.i31.9253.
[2]Carethers JM.Differentiating Lynch-like from Lynch syndrome[J].Gastroenterology, 2014, 146(3):602-604.doi: 10.1053/j.gastro.2014.01.041.
[3]Poulogiannis G, Frayling IM, Arends MJ.DNA mismatch repair deficiency in sporadic colorectal cancer and Lynch syndrome[J].Histopathology, 2010, 56(2):167-179.doi: 10.1111/j.1365-2559.2009.03392.x.
[4]Liu W, Zhang D, Tan SA, et al.Sigmoid Colon Adenocarcinoma with Isolated Loss of PMS2 Presenting in a Patient with Synchronous Prostate Cancer with Intact MMR: Diagnosis and Analysis of the Family Pedigree[J].Anticancer Res, 2018,38(8):4847-4852.doi: 10.21873/anticanres.12796.
[5]Peltomäki P, Gao X, Mecklin JP.Genotype and phenotype in hereditary nonpolyposis colon cancer: a study of families with different vs.shared predisposing mutations[J].Fam Cancer, 2001, 1(1):9-15.
[6]于鹏飞, 顾国利, 魏学明, 等.hMLH1、hMSH2、hMSH6在散发性结直肠癌中的表达状态与其临床病理特征的关系[J].中国普外基础与临床杂志, 2015, 22(5):581-585.doi: 10.7507/1007-9424.20150153.Yu PF, Gu GL, Wei XM, et al.Expression of hMLH1, hMSH2 or hMSH6 in Sporadic Colorectal Cancer and Relationship of It's Expression to Clinicopathologic Parameters[J].Chinese Journal of Bases and Clinics In General Surgery, 2015, 22(5):581-585.doi:10.7507/1007-9424.20150153.
[7]Stadler ZK.Diagnosis and management of DNA mismatch repairdeficient colorectal cancer[J].Hematol Oncol Clin North Am, 2015,29(1):29-41.doi: 10.1016/j.hoc.2014.09.008.
[8]Dray BK, Raveendran M, Harris RA, et al.Mismatch repair gene mutations lead to lynch syndrome colorectal cancer in rhesus macaques[J].Genes Cancer, 2018, 9(3/4):142-152.doi: 10.18632/genesandcancer.170.
[9]晋龙, 眭玉霞, 王丽萍, 等.结直肠癌错配修复蛋白MLH1、MSH2、MSH6及PMS2表达与临床病理特征的关系[J].诊断病理学杂志, 2017, 24(11):813-817.doi:10.3969/j.issn.1007-8096.2017.11.004.Jin L, Sui YX, Wang LP, et al.Correlation of the expression of mismatch repair proteins (MLH1, MSH2, MSH6 and PMS2)and clinicopathological parameters in colorectal carcinoma[J].Chinese Journal of Diagnostic Pathology, 2017, 24(11):813-817.doi:10.3969/j.issn.1007-8096.2017.11.004.
[10]Nicolaides NC, Papadopoulos N, Liu B, et al.Mutations of two PMS homologues in hereditary nonpolyposis colon cancer[J].Nature, 1994, 371(6492):75-80.doi:10.1038/371075a0.
[11]Wu J, Gu L, Wang H, et al.Mismatch repair processing of carcinogen-DNA adducts triggers apoptosis[J].Mol Cell Biol, 1999,19(12):8292-8301.doi:10.1128/mcb.19.12.8292.
[12]Gong Q, Zhang HH, Sun SB, et al.Mismatch repair-deficient status associates with favorable prognosis of Eastern Chinese population with sporadic colorectal cancer[J].Oncol Lett, 2018, 15(5):7007-7013.doi: 10.3892/ol.2018.8192.
[13]Li FY, Lai MD.Colorectal cancer, one entity or three[J].J Zhejiang Univ Sci B, 2009, 10(3):219-229.doi: 10.1631/jzus.B0820273.
[14]Kawakami H, Zaanan A, Sinicrope FA.Microsatellite instability testing and its role in the management of colorectal cancer[J].Curr Treat Options Oncol, 2015, 16(7):30.doi: 10.1007/s11864-015-0348-2.
[15]Yan WY, Hu J, Xie L, et al.Prediction of biological behavior and prognosis of colorectal cancer patients by tumor MSI/MMR in the Chinese population[J].Onco Targets Ther, 2016, 9:7415-7424.doi:10.2147/OTT.S117089.
[16]Gupta R, Sinha S, Paul RN.The impact of microsatellite stability status in colorectal cancer[J].Curr Probl Cancer, 2018, 42(6):548-559.doi: 10.1016/j.currproblcancer.2018.06.010.
[17]Laporte GA, Leguisamo NM, Kalil AN, et al.Clinical importance of DNA repair in sporadic colorectal cancer[J].Crit Rev Oncol Hematol, 2018, 126:168-185.doi:10.1016/j.critrevonc.2018.03.017.
[18]Ten Broeke SW, van Bavel TC, Jansen AML, et al.Molecular Background of Colorectal Tumors From Patients With Lynch Syndrome Associated With Germline Variants in PMS2[J].Gastroenterology, 2018, 155(3):844-851.doi:10.1053/j.gastro.2018.05.020.
[19]Kim ST, Klempner SJ, Park SH, et al.Correlating programmed death ligand 1 (PD-L1) expression, mismatch repair deficiency, and outcomes across tumor types: implications for immunotherapy[J].Oncotarget, 2017, 8(44):77415-77423.doi:10.18632/oncotarget.20492.
[20]Liu S, Kong P, Wang X, et al.Tumor microenvironment classification based on T-cell infiltration and PD-L1 in patients with mismatch repair-proficient and -deficient colorectal cancer[J].Oncol Lett, 2019, 17(2):2335-2343.doi: 10.3892/ol.2018.9826.
